ColabFold
ColabFold 为 Sergey Ovchinnikov 等人开发的适用于 Google Colab 的 AlphaFold 版本,使用 MMseqs2 替代 Jackhmmer,且不使用模版。ColaFold 计算迅速,短序列五六分钟即可算完。
注意事项:
- 运行 ColabFold 作业需要丰富的GPU资源,建议选择通用二区 V100 单卡进行计算。
- 输入文件名不能有特殊符号和
空格。 - 模板提交一次上传多个序列文件,会启动多个计算节点,每个计算节点对应一个序列文件。
- 需要注意提交的序列文件格式,详情请看以下示例:
单体结构输入文件示例:
>sequence_1
GSDNGFGSSKATSGSDFGGLAIFDGSGSEHFGHSDTHGSFDGLFGVDFZILSQQLKS
如果是多聚体结构,序列之间用分号隔开,输入文件示例:
>sequence_1
FADGAFGSGSDFSGFHGFGGHSRADGGTGFDGDF:LQFDSAFQLKSKFDOASFJAPOFJDPAISFJPAFJKAPF
一. 模板提交
ColabFold 使用模板提交,会根据上传的输入文件个数,启动对应数量的作业节点。如:上传5个输入文件,硬件配置选择节点数量为1提交作业,默认会启动5个作业节点同时计算,相应的以5个节点的价格进行计费;相比于串行提交,以相同的成本节约了计算的时间。
Step 1. 在应用中心搜索ColabFold软件,点击提交作业-选择模板提交;

Step 2.选择可视化模板提交;

Step 3. 上传序列文件,选择运行模式(单体选择monomer,多聚体选择multimer);

Step 4. 选择GPU硬件配置虎鲸1卡(v100-1);
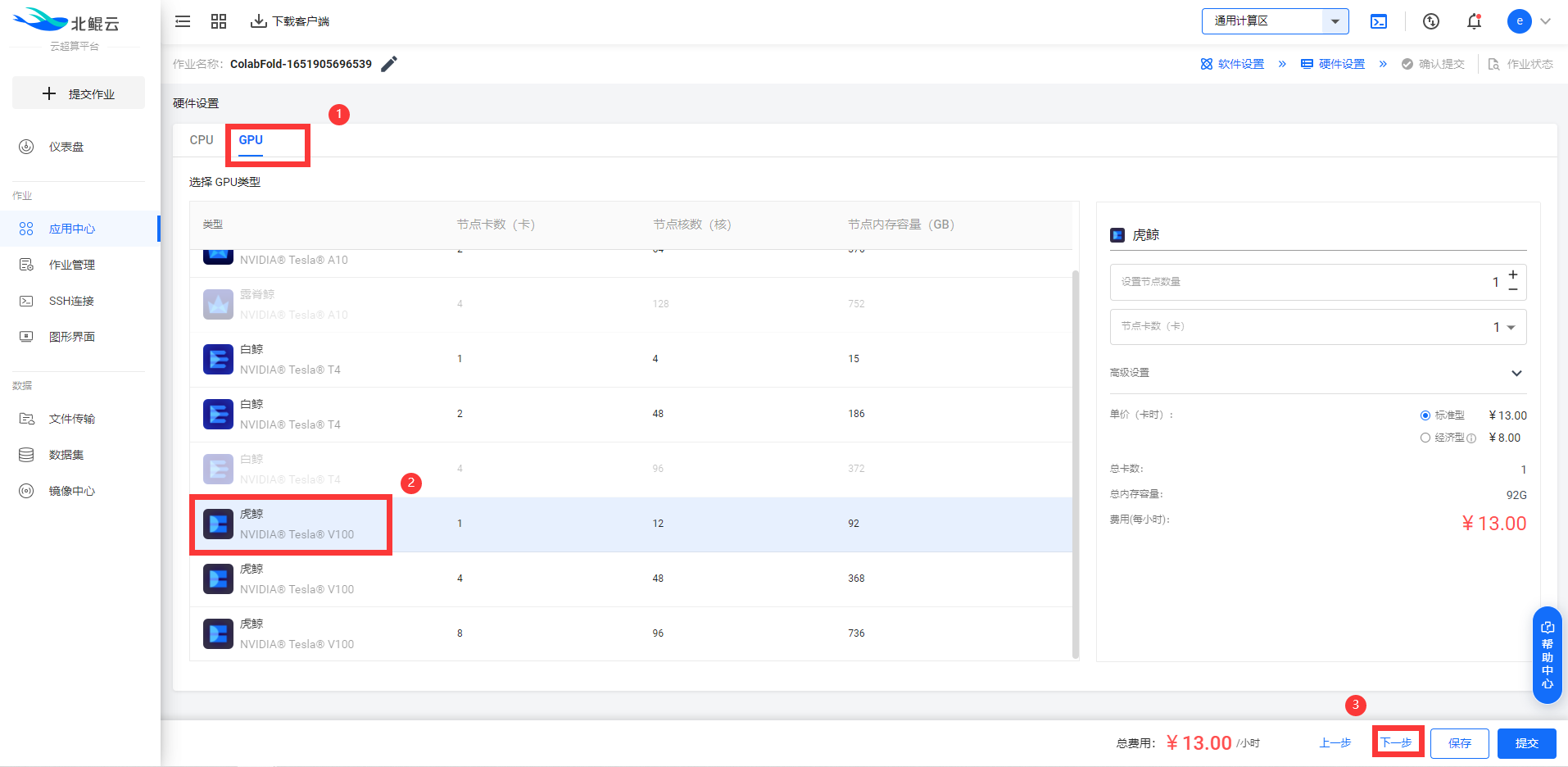
Step 5. 查看作业内容汇总,并提交作业;

Step 6. 通过作业管理查看运行中的作业;
二. 命令行提交
通过SSH连接创建并连接管理节点。
CPU版 ColabFold 作业示例
Step 1. 创建作业目录并进入;
mkdir colabfoldJob1
cd colabfoldJob1
Step 2. 通过文件传输上传所需的输入文件test.fasta,详情请查看Linux数据传输;
Step 3. 在该文件夹下创建如下执行脚本colabfold.sh:
#!/bin/bash
module add ColabFold/1.5.2
#如果是预测单体
colabfold_batch --amber --templates --num-recycle 3 test.fasta output
#如果是预测多聚体
#colabfold_batch --amber --templates --num-recycle 3 --model-type alphafold2_multimer_v3 test.fasta output
Step 4. 提交作业;
提交任务到一台16核的CPU节点运行。
sbatch -N 1 -p c-16-2 -n 16 -c 1 colabfold.sh
GPU版 ColabFold 作业示例
Step 1. 创建作业目录并进入;
mkdir colabfoldJob1
cd colabfoldJob1
Step 2. 通过文件传输上传所需的输入文件test.fasta,详情请查看Linux数据传输;
Step 3. 在该文件夹下创建如下执行脚本colabfold.sh:
#!/bin/bash
module add ColabFold/1.5.2
#如果是预测单体
colabfold_batch --amber --templates --use-gpu-relax --num-recycle 3 test.fasta output
#如果是预测多聚体
#colabfold_batch --amber --templates --use-gpu-relax --num-recycle 3 --model-type alphafold2_multimer_v3 test.fasta output
Step 4. 提交作业;
提交任务到带有一张V100卡的GPU节点运行。
sbatch -p g-v100-1 -c 10 colabfold.sh
查看作业运行情况及参数详细介绍请点击查看slurm命令。
结果文件下载请查看Linux数据传输。