


上期四川大学李建宗博士为大家分享了《手把手教你用Gromacs完成溶菌酶在水中的动力学模拟》。
今天,将继续为大家介绍如何在北鲲云计算平台上利用Amber完成蛋白-小分子体系的动力学模拟,以及利用MMGBSA计算小分子与蛋白质(Abl和伊马替尼)之间的结合自由能。
Amber是美国加州大学Peter Kollman等开发的一款著名的分子动力学模拟软件包。Amber主要适用于蛋白质,小分子和多糖等生物分子体系的模拟。
01 结构处理
建议现在自己电脑上安装一个UCSF Chimera小程序,用于处理分子模拟所需的结构文件。该软件学术免费,而且具备和PyMOL类似的图形显示功能。获取地址:
https://www.cgl.ucsf.edu/chimera/download.html
获取晶体结构结构复合物
本教程旨在利用Amber在北鲲云平台上完成蛋白质Abl和靶向药物伊马替尼体系的动力学模拟,并且计算他们之间的结合自由能。幸运的是PDB数据库中包含了Abl和伊马替尼的复合物的晶体结构,编号为6E4F。通过UCSF Chimera的fetch工具可以方便的获取晶体结构。在File—Fetch by ID中的PDB后输入编号6E4F即可下载复合物晶体结构,并将其保存为6E4F.pdb到自定义工作路径中。
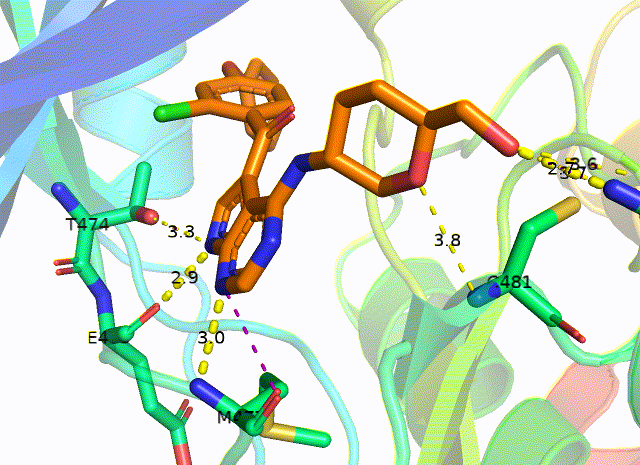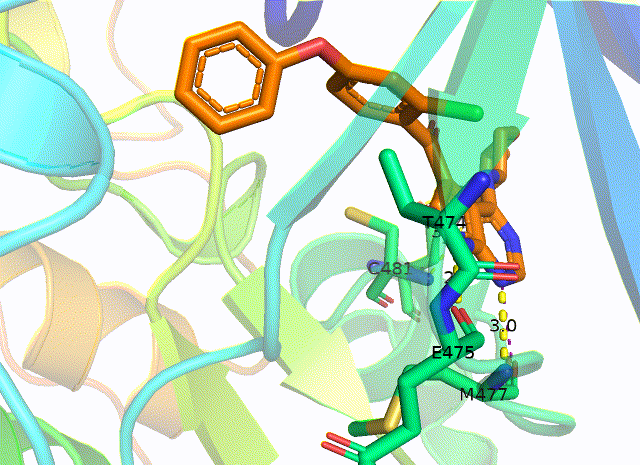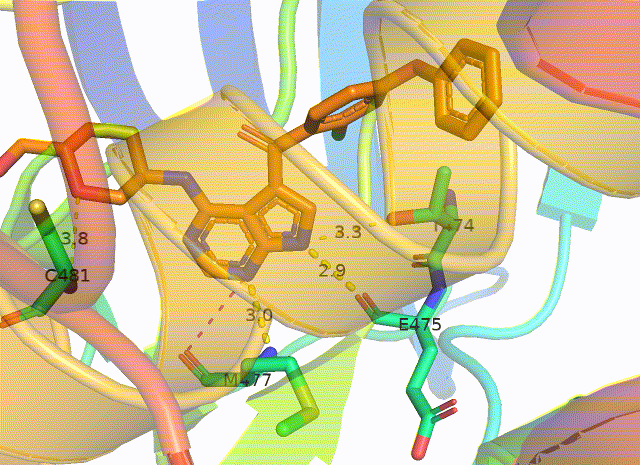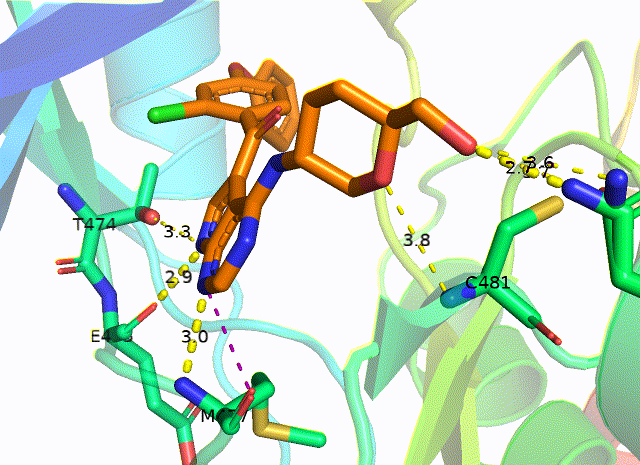
Abl和伊马替尼的相互作用细节
处理蛋白质
下载复合物晶体结构以后,先对蛋白质进行处理,删除复合物中所有的除开蛋白质外的原子。在Residue中选择所有非标准残基,然后通过Actions—Atoms/bonds—delete进行删除。将蛋白质保存为protein.pdb文件。
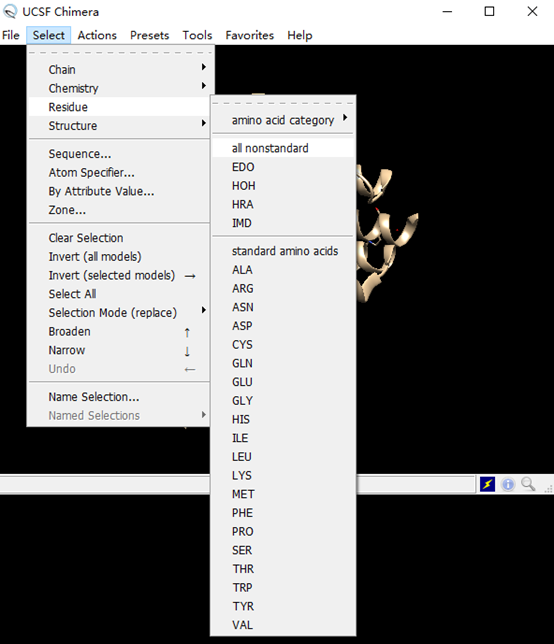
处理配体
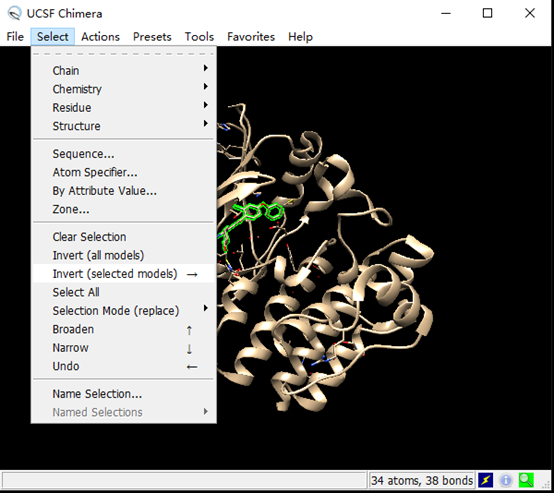
复合物中伊马替尼的名称为HRA,重新打开6E4F.pdb文件,然后选中HRA,在Select菜单中用Invert选中其余分子,并且删除,只保留伊马替尼的结构。如下所示。
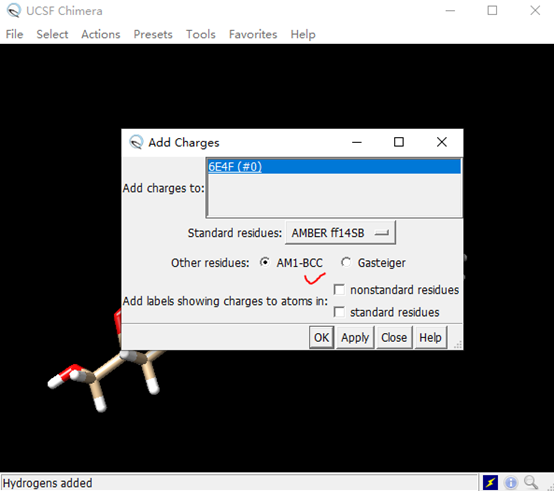
然后在Tools中找到Structure Editing分别对小分子进行加氢(Add H)和加电荷(Add charge)处理,电荷选择AM1-BCC。并将处理后的小分子保存为ligand.mol2文件。
02 上传输入文件到北鲲云计算平台
登录北鲲云计算平台,将处理好的结构文件上传至北鲲云计算平台,然后在提交作业中选择图形界面提交或者命令界面提交。北鲲云可快速帮你安装诸如Amber在内的模拟软件,免去了安装和环境配置等繁琐步骤,而且上传输入文件和下载计算结果文件都是图形操作,直观且方便。
Amber支持GPU加速,在NVIDIA GPU 上的运行速度比仅使用 CPU 的系统快 15 倍,因此,在提交作业的时候注意选择GPU计算区,这里的GPU计算资源十分丰富。对于不需要GPU的作业,可以选择通用算区节省成本。
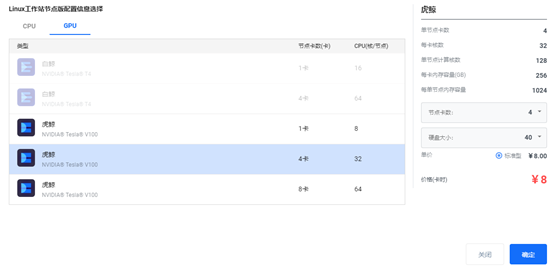
通过图形界面提交任务,然后启动计算节点后,进入到我们刚才上传的文件夹下,可以看到如下所示的操作系统界面,北鲲云计算平台目前使用的是Centos 7系统,鼠标右键选择Open in Terminal然后就可以开始进行动力学模拟和结合自由能计算了。
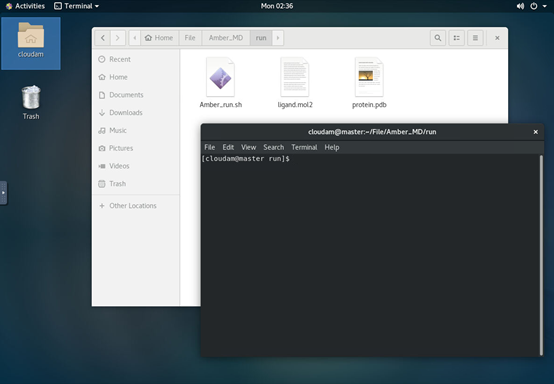
03 动力学模拟
本教程展示在北鲲云计算平台上进行动力学模拟的具体细节,计算量大的任务请参考本教程第04小结进行任务提交。
利用Antechamber处理配体,并且利用parmchk2检查转换为Amber识别的参数结构文是否正确,命令如下:
antechamber -i ligand.mol2 -fi mol2 -o ligand.ante.mol2 -fo mol2 parmchk2 -i ligand.ante.mol2 -f mol2 -o ligand.frcmod
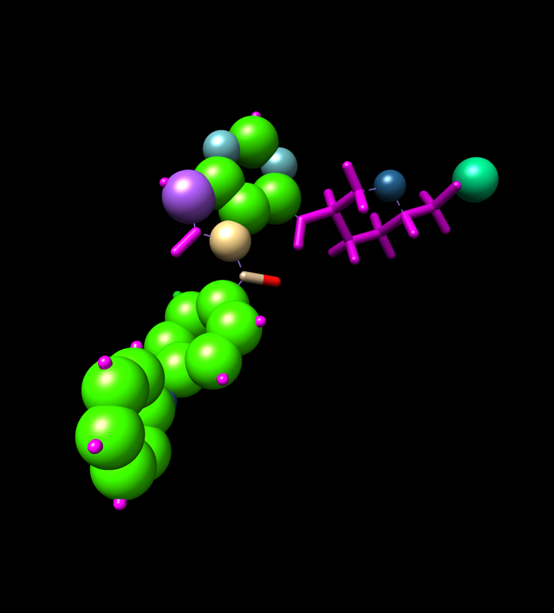
其中-i指定输入文件,-fo或则-f指定输入文件格式,-o指定输出文件,-fo指定输出文件格式。检查是否有ligand.ante.mol2和ligand.frcmod文件生成。利用Chimera打开ligand.ante.mol2,可观测到如下参数化后的结构。
然后利用tleap模块生成配体、蛋白以及复合物的拓扑文件和模拟初始文件。首先调用tleap
tleap
然后在tleap窗口中分别加载小分子、蛋白质和溶剂所需要的力场参数
source leaprc.protein.ff14SBsource leaprc.gaffsource leaprc.water.tip3p
其中protein.14SB指定蛋白质力场,gaff指定小分子力场,tip3p指定水分子模型。
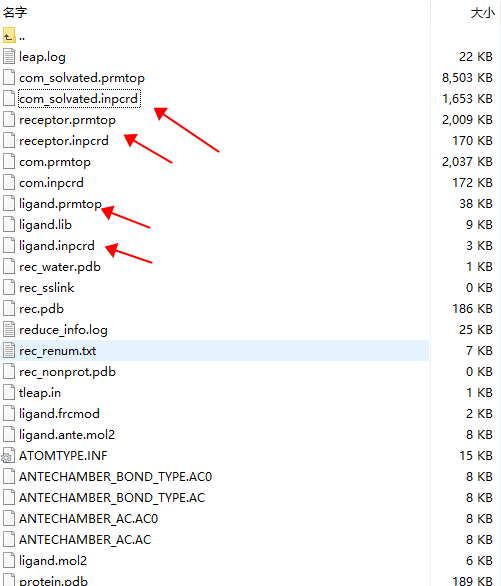
然后生成配体初始文件ligand.prmtop和ligand.inpcrd,并且检测配体结构完整性。
LIG = loadmol2 ligand.ante.mol2check LIGloadamberparams ligand.frcmodsaveoff LIG ligand.libsaveamberparm LIG ligand.prmtop ligand.inpcrd
生成蛋白初始文件receptor.prmtop和receptor.inpcrd
REC = loadpdb rec.pdbcheck RECsaveamberparm REC receptor.prmtopreceptor.inpcrd
生成蛋白-配体复合物初始文件com_solvated.prmtop和com_solvated.inpcrd
COM = combine {REC, LIG}saveamberparm COM com.prmtop com.inpcrdcharge COMsolvatebox COM TIP3PBOX 12.0saveamberparm COM com_solvated.prmtopcom_solvated.inpcrdtleap会有类似提示
Building topology.Building atom parameters.Building bond parameters.Building angle parameters.Building proper torsion parameters.Building improper torsion parameters.total943 improper torsions appliedBuilding H-Bond parameters.Incorporating Non-Bonded adjustments.Not Marking per-residue atom chain types.Marking per-residue atom chain types.(Residues lacking connect0/connect1 -these don't have chain types marked:res total affected
完成上述操作后,没有错误提示,则可退出tleap
quit
如果成功生成下面这些这些文件,则可以开始下一步的能量优化了。
能量最小化
动力学模拟起始需要对体系进行初始能量最小化,以消除不合理原子间的碰撞和不规范的作用力,命令如下:
pmemd.cuda -O -i min.in -o min.out -p com_solvated.prmtop -c com_solvated.inpcrd -r min.rst -ref com_solvated.inpcrd
pmemd.cuda,调用pmemd的GPU版本,这里-i指定参数文件min.in, 对体系的平衡进行了限定,参考官网指南具体min.in内容
minimise com&cntrlimin=1,maxcyc=2000,ncyc=1000,cut=8.0,ntb=1,ntc=2,ntf=2,ntpr=100,ntr=1, restraintmask=':1-288',restraint_wt=2.0/
imin=1 指定模拟步骤为能量最小化
maxcyc表明采用共轭梯度算法,且指定最大循环数,
ncyc=1000表明采用最陡下降算法
ntpr=100表明保存模拟信息间隔
restraintmask指定限制氨基酸范围
restraint_wt=2.0表示限制力的大小
升温模拟
对体系进行升温,温度从0K开始到300K结束,命令如下,并且检测是否有heat.rst和 heat.mdcrd文件生成。
pmemd.cuda -O -i heat.in -o heat.out -p com_solvated.prmtop -c min.rst -r heat.rst -x heat.mdcrd -ref min.rst
heat.in参数内容
heat ras-raf&cntrlimin=0,irest=0,ntx=1,nstlim=25000,dt=0.002,ntc=2,ntf=2,cut=8.0, ntb=1,ntpr=500, ntwx=500,ntt=3, gamma_ln=2.0,tempi=0.0, temp0=300.0, ig=-1,ntr=1, restraintmask=':1-242',restraint_wt=2.0,nmropt=1/&wt TYPE='TEMP0', istep1=0, istep2=25000,value1=0.1, value2=300.0, /&wt TYPE='END' /
密度平衡
对体系进行密度平衡模拟,检查是否有density.rst和density.mdcrd文件生成 ,命令如下:
pmemd.cuda -O -i density.in -o density.out -p com_solvated.prmtop -c heat.rst -r density.rst -x density.mdcrd -ref heat.rst
所用参数文件 density.in,
heat ras-raf&cntrlimin=0,irest=1,ntx=5,nstlim=25000,dt=0.002,ntc=2,ntf=2,cut=8.0, ntb=2, ntp=1, taup=1.0,ntpr=500, ntwx=500,ntt=3, gamma_ln=2.0,temp0=300.0, ig=-1,ntr=1, restraintmask=':1-242',restraint_wt=2.0,/
体系平衡
最后进行体系平衡模拟,检查是否有equil.rst和equil.mdcrd生成
pmemd.cuda -O -i equil.in -o equil.out -p com_solvated.prmtop -c density.rst -r equil.rst -x equil.mdcrd
equil.in参数文件内容如下:
heat ras-raf&cntrlimin=0,irest=1,ntx=5,nstlim=250000,dt=0.002,ntc=2,ntf=2,cut=8.0, ntb=2, ntp=1, taup=2.0,ntpr=1000, ntwx=1000,ntt=3, gamma_ln=2.0,temp0=300.0, ig=-1,/
利用process_mdout.pl脚本检查上述平衡是否达到合理水平,命令如下
process_mdout.pl heat.out density.out equil.out
利用xmgrace作图:
xmgrace summary.DENSITYxmgrace summary.TEMPxmgrace summary.ETOT
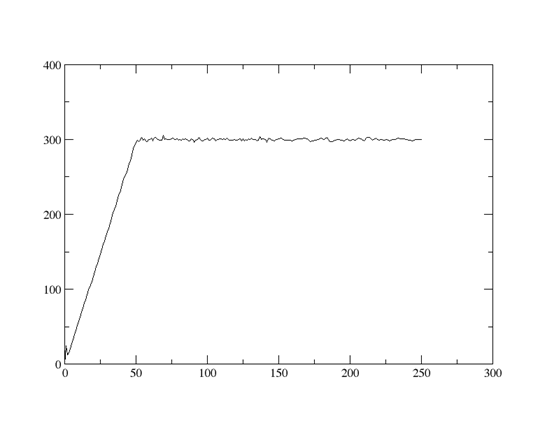
温度平衡结果
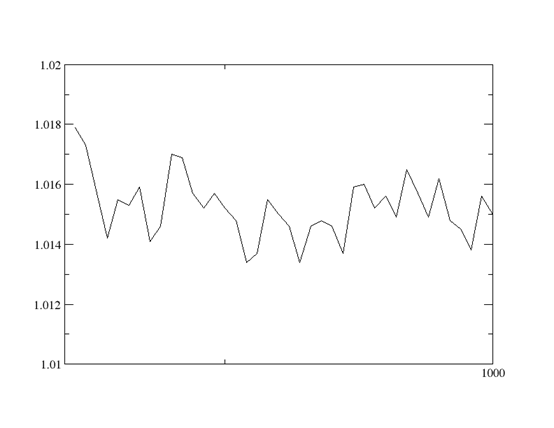
密度平衡结果
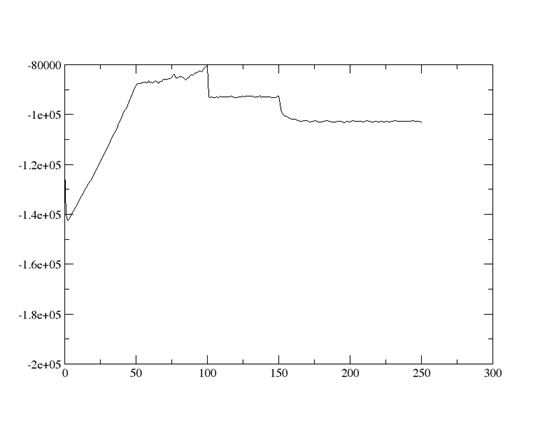
体系平衡结果
成品模拟
从上面分析可知体系基本上达到平衡,因此可以进行成品模拟步骤,命令如下
pmemd.cuda -O -i prod.in -o prod.out -p com_solvated.prmtop -c equil.rst -r prod1.rst -x prod1.mdcrd
prod.in参数为:
heat ras-raf&cntrlimin=0,irest=1,ntx=5,nstlim=500000,dt=0.002,ntc=2,ntf=2,cut=8.0, ntb=2, ntp=1, taup=2.0,ntpr=5000, ntwx=5000,ntt=3, gamma_ln=2.0,temp0=300.0, ig=-1, /
同样的利用Chimera的MD movie工具打开模拟结果文件的prod1.mdcrd和com_solvated.prmtop,观看动力学模拟动画效果。
Amber动力学模拟动态展示效果
通过MD movie--Analysis--Plot--RMSD工具绘制体系的RMSD图,分析结果表明整个体系在模拟的时间段内处于2.0 Å范围内波动,是合理范围,可用于后续结合自由能计算。
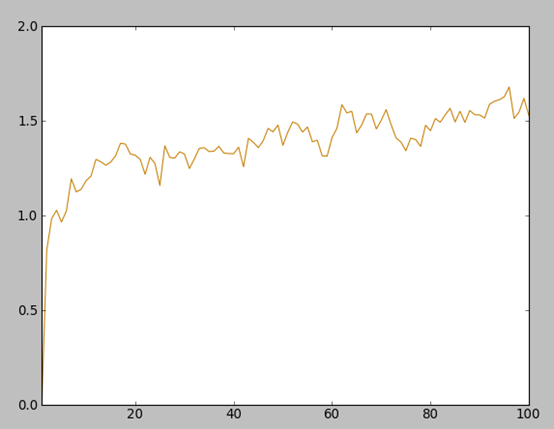
动力学模拟体系RMSD结果
04 结合自由能计算
Amber集成了很多种结合自由能计算,例如MMGBSA或者MMPBSA,这里我们用MMGBSA来计算Abl和伊马替尼的结合自由能,Amber中计算结合自由能的模块为MMPBSA.py命令如下:
python $AMBERHOME/bin/MMPBSA.py -O -i mmpbsa.in -o result.dat -sp com_solvated.prmtop -cp com.prmtop -rp receptor.prmtop -lp ligand.prmtop -y prod*.mdcrd
mmpbsa.in参数文件指定了计算结合自由能类型,具体参数内容如下:
Input file for running PB and GB&general startframe=5, verbose=1,# entropy=1,/&gb igb=2, saltcon=0.100/&pb istrng=0.100,/
运行完毕会生成一系列记载能量信息的文件,最终结果包含在命令指定的dat后缀的文件中,可提取数值进行作图分析。通过计算可知Abl蛋白质与imatinib的结合自由能为-62.73 kcal/mol,具体能量项如下图所示,主要包括了范德华相互作用、静电相互作用、溶剂自由能等。
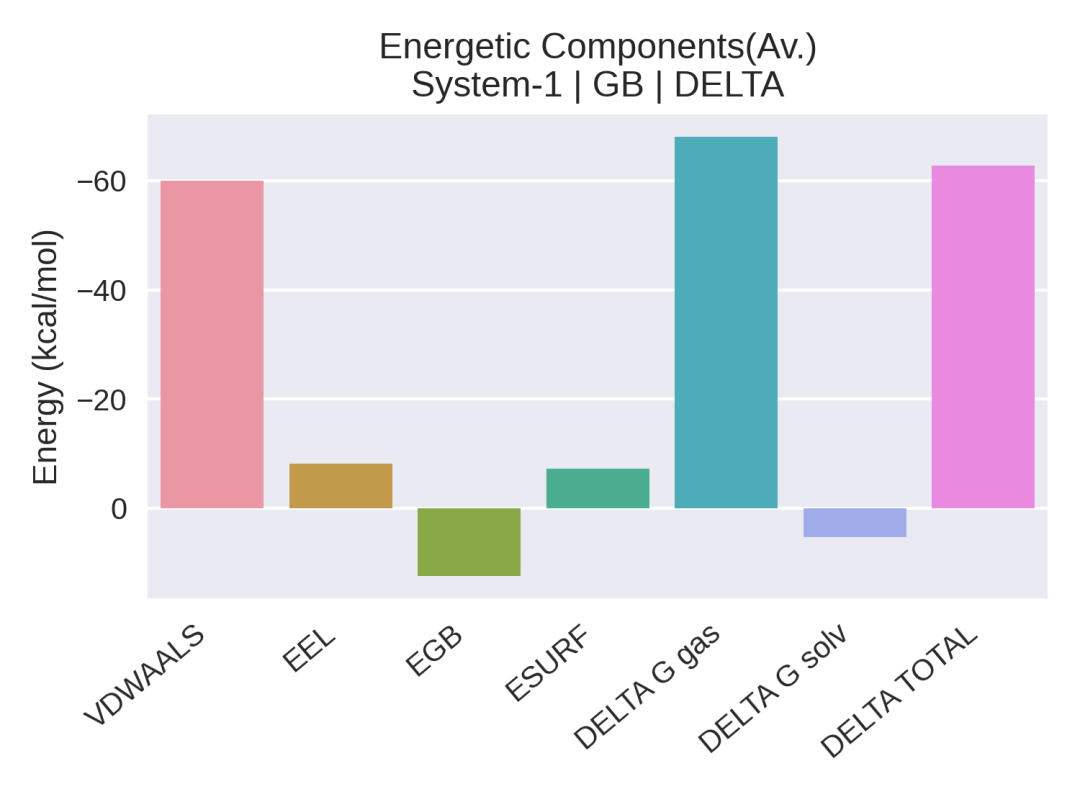
Abl和imatinib结合自由能计算结果
05 北鲲云计算平台作业提交
上述教程是在北鲲云计算平台上运行的细节,在计算平台上提交作业,请不要在管理节点逐条需要消耗计算资源的命令,管理节点只有2核4G内存的计算资源,进行动力学模拟必须将作业提交到运算节点进行。
因此需要将上述命令写进脚本中,然后利用如下命令提交即可:
sbatch -N 1 -p g-v100-1 -c 12 amber.sh
N为节点的数量,这里输入的是1。p为选择的PARTITION,这里使用的是V100卡(g-v100-1)。查看作业运行情况及参数详细介绍可使用slurm命令进行查看。
amber.sh内容是上述教程中所有命令的脚本形式,内容为:
#!/bin/bashmodule add Anaconda3/2020.02source /public/software/.local/easybuild/software/amber/aber20/amber.shulimit -s unlimitedulimit -l unlimitedantechamber -i ligand.mol2 -fi mol2 -o ligand.ante.mol2 -fo mol2parmchk2 -i ligand.ante.mol2 -f mol2 -o ligand.frcmodpdb4amber -i protein.pdb -o rec.pdb --reduce --dry -y --nohydtleap -s -f tleap.inpmemd.cuda -O -i min.in -o min.out -p com_solvated.prmtop -c com_solvated.inpcrd -r min.rst -ref com_solvated.inpcrdpmemd.cuda -O -i heat.in -o heat.out -p com_solvated.prmtop -c min.rst -r heat.rst -x heat.mdcrd -ref min.rstpmemd.cuda -O -i density.in -o density.out -p com_solvated.prmtop -c heat.rst -r density.rst -x density.mdcrd -ref heat.rstpmemd.cuda -O -i equil.in -o equil.out -p com_solvated.prmtop -c density.rst -r equil.rst -x equil.mdcrdpmemd.cuda -O -i prod.in -o prod1.out -p com_solvated.prmtop -c equil.rst -r prod1.rst -x prod1.mdcrdpython $AMBERHOME/bin/MMPBSA.py -O -i mmpbsa.in -o result.dat -sp com_solvated.prmtop -cp com.prmtop -rp receptor.prmtop -lp ligand.prmtop -y prod*.mdcrd
上述脚本中前面5行命令是告诉北鲲云计算平台需要调用Amber程序,后面的命令就是本教程所使用的具体命令了。
因此只需要将protein.pdb和ligand.mol2文件、所有in参数文件上传到北鲲云计算平台,然后提交amber.sh作业脚本即可完成本教程所有内容。本教程参数文件获取地址:
https://pan.baidu.com/s/1n3HuK9dp9o_uTdBjLuPpdQ 提取码: 49x8
高性能计算资源,极大的节省计算成本
支持海量的计算工具,且开箱即用
计算任务进度实时追踪提示的贴心服务
详细耐心的技术咨询